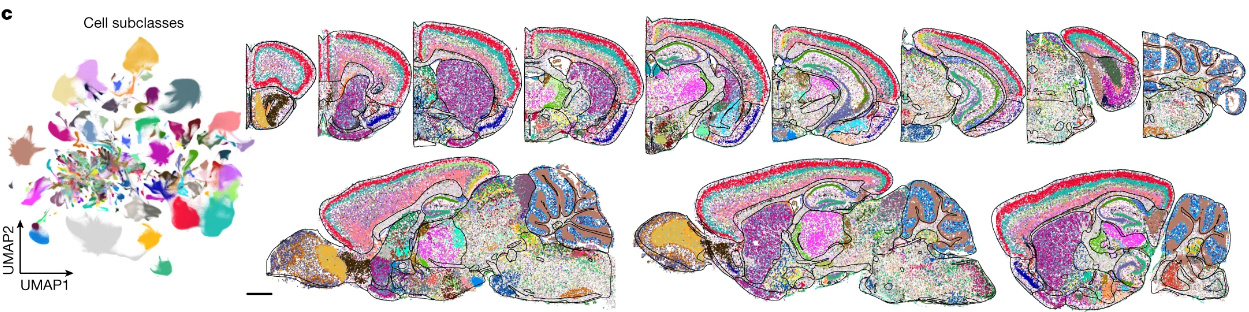
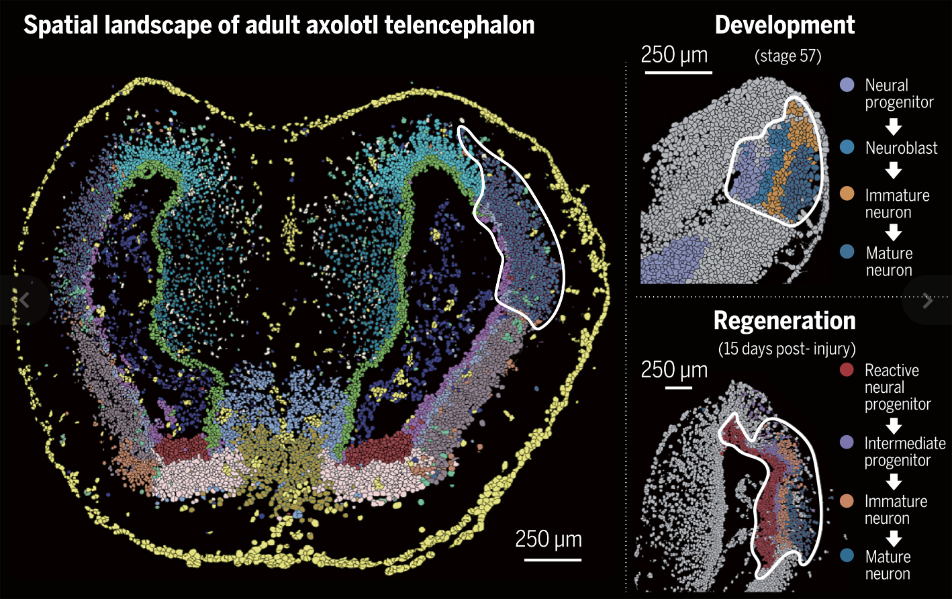
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81
82
83
84
85
86
87
88
89
90
91
92
93
94
95
96
97
98
99
100
| import scanpy as sc
import pandas as pd
import anndata as ad
from scipy.sparse import csr_matrix
import numpy as np
import numpy as np
from PIL import Image
cellbin = ad.read('/data/work/07.3d/Result/Spateo/0112/slices/20.h5ad')
'''
AnnData object with n_obs × n_vars = 19415 × 17887
obs: 'area', 'x', 'y', 'region', 'n_counts', 'slices', 'batch', 'celltype', 'celltype_main', 'celltype_main_sub', 'z'
uns: 'celltype_colors', 'celltype_main_colors', 'celltype_main_sub_colors', 'data_unit', 'region_colors'
obsm: '3d_align_spatial', '3d_align_spatial_confine', 'Nonrigid_align_spatial', 'Rigid_align_spatial', 'X_umap', 'align_spatial', 'bbox', 'spatial'
'''
cellbin.obs.index = [i.split('-')[0] for i in cellbin.obs.index.tolist()]
adata = ad.read('/data/work/01.Cellbin/02.Result/20/0804/Slice_20_labeled.h5ad')
'''
AnnData object with n_obs × n_vars = 14162 × 15326
uns: '__type', 'pp', 'spatial'
layers: 'augment_labels', 'augmented_labels_expanded', 'stain', 'stain_distances', 'stain_markers', 'stain_mask', 'watershed_labels'
'''
image = adata.layers['augmented_labels_expanded'].copy()
del adata
uni = np.unique(image)
image_g = []
for i in uni:
if str(i) in cellbin.obs.index:
continue
else:
image_g.append(i)
control = np.isin(image, image_g)
control = ~control
image = np.where(control, image, 0)
new_uni = np.unique(image)
dic = {}
k = max(new_uni)+1
for i in set(cellbin.obs['celltype']):
dic[i] = k
k+=1
index = [int(i) for i in cellbin.obs.index.tolist()]
color = list(set(cellbin.uns['celltype_colors']))
def Hex_to_RGB(hex):
r = int(hex[1:3],16)
g = int(hex[3:5],16)
b = int(hex[5:7], 16)
rgb = (r,g,b)
return rgb
color_dict = {
0: (0, 0, 0),
}
for i,index in enumerate(dic):
color_dict[dic[index]] = Hex_to_RGB(color[i])
for i in cellbin.obs.index:
region = cellbin.obs.loc[i].celltype
reigon_id = dic[region]
image[image==int(i)] = reigon_id
height, width = image.shape
image_data = np.zeros((height, width, 3), dtype=np.uint8)
for value, color in color_dict.items():
image_data[image == value] = color
image_save = Image.fromarray(image_data)
image_save.save('/data/work/07.3d/Script/0116/2.png')
|